13. Dimensionality Reduction
- -
Curse of Dimensionality
Dimensional Reduction
기존 데이터들의 properties들을 보존하면서 high-dimensional space를 low-dimensional space로 내리고자 하는것
→ It is commonly used for
- feature extraction
- data compression
- data visualization
Linear dimensionality reduction
- Principal component analysis (PCA)
- Factor analysis
Non-linear dimensionality reduction
- Kernel principal component analysis (Kernel PCA)
- T-SNE
Linear Dimensionality Reduction
Data Projection into Subspace

We need to find a new basis of 2D space that can carry most of the information
→ 그렇다면 어떤 기준으로 basis를 정해야하는 것인가?
이에 대한 기준 2가지를 순서대로 살펴보도록 하자.
Criteria 1 : Maximum Variance Formulation
Given a dataset {xn}n=1N where xn∈RD, the goal is to project the data onto a space having dimensionality M<D while maximizing the variance of the projected data.

첫 번째 기준은 data들의 variance가 가장 크게끔 basis를 설정하는 것이다. 구체적인 상황을 1차원에서 확인하고, 이를 다차원으로 확장시켜보도록 하자.
1-dimensional Principal Subspace
xn을 u에 projection 시키고자 하는 것 uTxn∈R1
The variance of the projected data is given as
이때, S를 Empirical covariance matrix of the data 라고 부른다.
Proof
We are asked to prove that
N1n=1∑N(uTxn−uTxˉ)2=uTSuwhere xn is the n-th data point in the dataset, xˉ is the mean of the data set, u is a vector, and S is the covariance matrix of the data set.
To start, expand the square in the left-hand side (LHS) of the equation:
N1n=1∑N(uTxn−uTxˉ)2=N1n=1∑N(uTxn)2−2(uTxn)(uTxˉ)+(uTxˉ)2=N1n=1∑N(uTxn)2−2(uTxˉ)N1n=1∑N(uTxn)+N1n=1∑N(uTxˉ)2=N1n=1∑N(uTxn)2−2(uTxˉ)(uTxˉ)+(uTxˉ)2=N1n=1∑N(uTxn)2−(uTxˉ)2Now, let's work on the right-hand side (RHS) of the equation:
uTSu=uT(N1n=1∑N(xn−xˉ)(xn−xˉ)T)u=uT(N1n=1∑NxnxnT−N1n=1∑NxnxˉT−N1n=1∑NxˉxnT+N1n=1∑NxˉxˉT)u=N1n=1∑NuTxnxnTu−N1n=1∑NuTxnxˉTu−N1n=1∑NuTxˉxnTu+N1n=1∑NuTxˉxˉTu=N1n=1∑N(uTxn)2−N1n=1∑N(uTxn)(uTxˉ)−N1n=1∑N(uTxˉ)(uTxn)+N1n=1∑N(uTxˉ)2=N1n=1∑N(uTxn)2−2(uTxˉ)(uTxˉ)+(uTxˉ)2As you can see, the LHS and the RHS are the same, which completes the proof.
PCA can be formulated as
Lagrange multiplier를 적용하면
즉 u must be the eigenvector of S having eigenvalueλ
따라서
M-dimensional Principal Subspace
이를 확장시켜서 basis를 M개 잡아보도록 하자.
Let {u1,…,uM} is a basis. PCA can be given as the maximization of total variance
→ 여기서 S∈MD×D
1차원에서와 동일하게 Lagrange multiplier를 처리하게 되면 다음과 같은 결론을 얻을 수 있다.
M개의 eigenvalue에 대응되는 eigenvector를 basis로 설정해주면 된다.Criteria 2 : Minimum Error Formulation
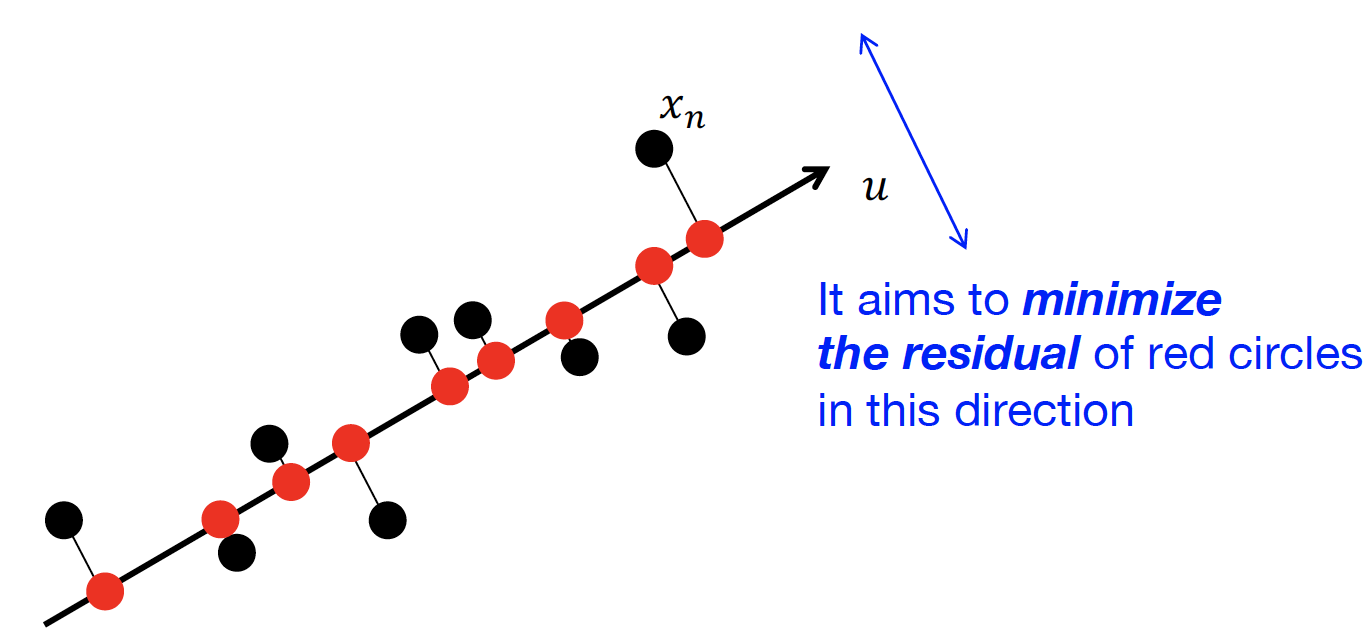
두 번째 기준은 정보의 손실을 최소로 하는 것이다. 즉 다음과 같은 projection error를 최소화하게끔하는 basis를 찾는 것이 목표이다.
→ 즉, projection matrix를 구하고 싶다고 이해해도 무방하다.
이때 우리는 다음과 같은 orthonormal basis를 잡았다고 가정하자.
(사실 Gram-schimdt를 활용하면 쉽게 orthonormal basis를 잡을 수 있다.)
그리고 일반성을 잃지 않고, 우리가 원하는 subspace의 basis를 {u1,…,uM}이라고 하자.
선형대수적인 지식을 활용하면 J를 작게 만들고 싶다는 의미는 결과적으로는 다음과 같이 정리할 수 있다.
그러면 다음과 같은 최적화 문제로 바뀌게 된다.
D - M개의 eigenvalue에 대응되는 eigenvector를 projection subspace의 basis로 설정해주면 된다.또한, 일반적으로 projection subspace의 basis를 principal component 라고 부른다.
PCA for High-Dimensional Data
일반적으로 dimension D에 비해서 data point의 개수 N이 작을 경우 dataset이 high-dimensional라고 부른다.
이때, empirical covariance matrix S의 eigenvalue decomposition을 하려면 결과적으로 O(D3)의 시간복잡도가 걸린다. (왜냐하면 S∈MD×D)
하지만, 잘 생각해보면 D 차원에 데이터가 N개가 있는 상황이므로, 데이터들이 형성할 수 있는 subspace는 기껏해봐야 N차원이다. 즉 dataset이 high-dimensional인 경우, S의 eigenvalue가 대부분 0이라는 것을 추측할 수 있다.
이러한 특성을 활용하면 data의 개수에 비해서 차원이 큰 경우에 computational cost를 줄일 수 있다.
일단 S를 다음과 같이 표현할 수 있고, 이를 활용하면 S의 eigenvalue를 구하는 과정을 다음과 같이 표현할 수 있다.
양변에 X를 곱해주면 다음과 같다.
즉 XXT의 eigenvector와 eigenvalue를 구하는 문제로 바뀌었다. 이때, 편의를 위해 Xui=vi라고 치환하자
XXT의 eigenvalue와 eigenvector를 구함으로써 vi를 구했다고 하자. 그렇다면 이거를 활용해서 ui로 어떻게 복원할 것인가?
위 식에 다시 XT를 곱해보자
이때, N1XTXui=λiui이므로
따라서
즉 이렇게 하면 시간복잡도를 O(N3)로 낮출 수 있다. 왜냐하면 결과적으로 XXT∈MN×N 이기 때문이다.
Factor Analysis

Factor analysis(FA) 및 probabilistic PCA (PPCA)는 generative model이다. 반면 앞에서 다룬 PCA는 데이터들이 놓여있는 차원보다 단순히 더 낮은 subspace로 projection시킨 것에 불과하다.
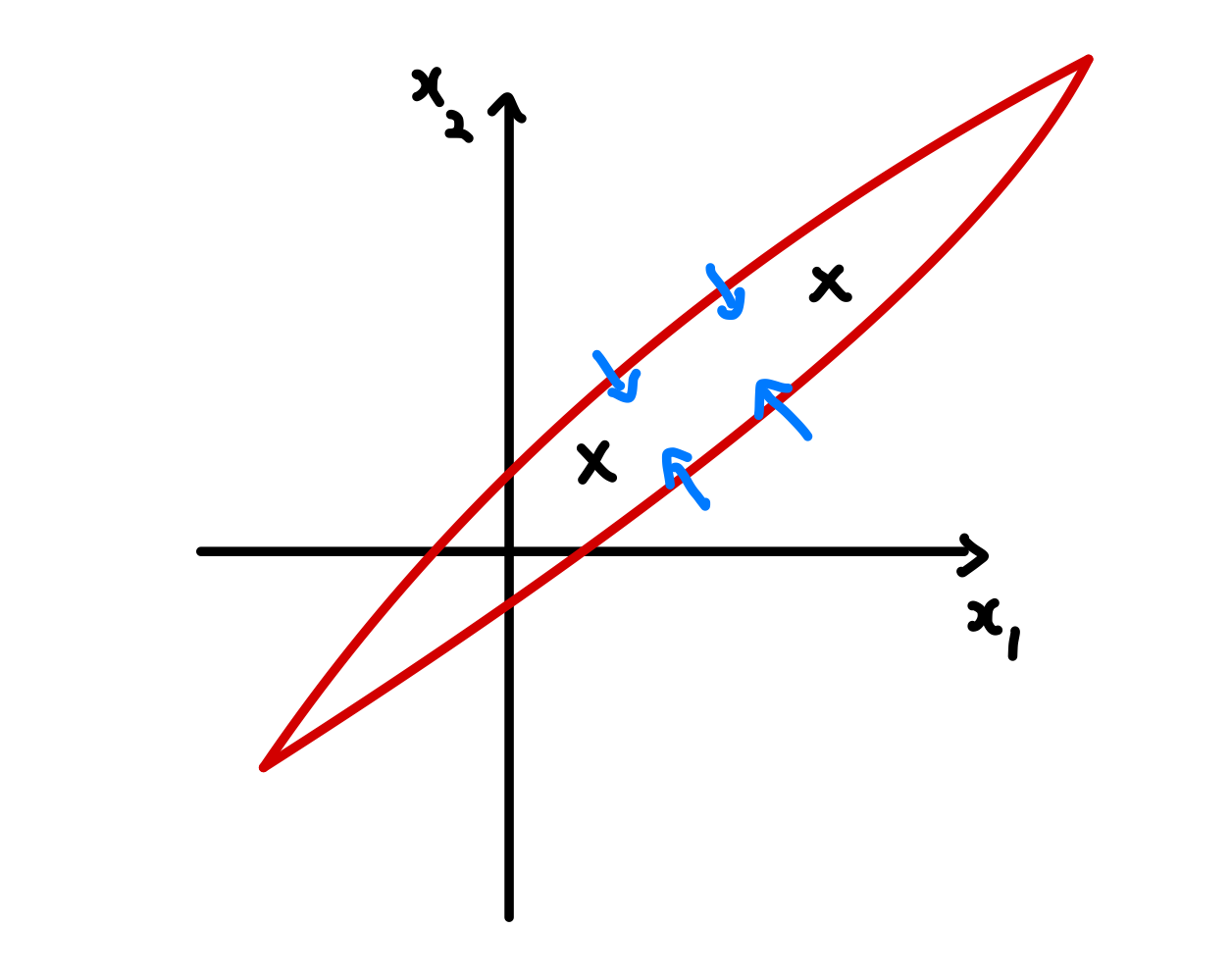
예를 들어 위 예시를 보면, 사람이라면 각 cluster에 대해 projection을 다르게 진행해야한다는 점을 판단할 수 있다. 하지만, PCA는 이러한 판단을 할 수 없다. 반면, factor analysis는 data의 distribution을 찾는 generative model이기 때문에 가능하다.
Revisit Mixture of Gaussian
Mixture of Gaussian의 경우 data가 놓여있는 차원보다 데이터의 수가 많이 작은 경우에 사용하기가 부적합하다.
예를 들어 x∼N(μ,Σ) 라고 가정하자. (단순히 1개의 Gaussian이라고 생각하자.)
이에 대한 MLE는 다음과 같을 것이다.
이때, 데이터들이 놓인 차원보다 데이터들의 개수가 더 적다고 가정하면 covariance matrix인 Σ 가 singular가 된다.
이렇게 되면 Multivariable Gaussian distribution을 계산하기가 어려워진다.
추가적으로 만약 2개의 2차원 상에 존재하는 데이터가 존재하고, Multivari-Gaussian 1개를 통해 데이터들의 분포를 추정하고 싶다고 가정하자. 그러면 Mixture of Gaussian에 의해서 추정되는 Gaussian은 굉장히 flat한 형태의 Gaussian이 될 것이다. 이러한 측면으로 봐도 covariance matrix가 singular가 된다는 것을 확인할 수 있다.
만약 여전히 Mixture of gaussian을 쓰고 싶은 경우에는 어떻게 해야할까? 정답은 Σ에 restriction 을 주면 된다.
Option 1 : Σ to be diagonal
Option 2 : Σ=σ2I
Option 2는 Option 1에 비해서 더 강한 가정을 하고 있다.
이렇게 되면 Gaussian는 Circular 형태를 띄게 된다. MLE를 통해 σ2를 esimate한 결과는 다음과 같다.
앞서 살펴본 것처럼, option 1 과 option 2는 feature들 사이의 관계를 무시하게 되는 문제점이 존재하게 된다. 이를 해결하기 위해서 등장한 개념이 factor analysis이다.
Definition of Factor analysis
Factor analysis is a statistical method used to describe variability among observed, correlated variables in terms of a potentially lower number of unobserved variables called factors. For example, it is possible that variations in six observed variables mainly reflect the variations in two unobserved (underlying) variables. The observed variables are modeled as linear combinations of the potential factors plus error terms
즉, 결과적으로 latent variable들의 linear combination과 error term들의 합으로 observed data를 표현하고 싶은 것이다.
Exact procedure
일단 다음과 같이 가정한다.
Diagonal) 따라서
Example 1
z∈R1,x∈R2(k=1,d=2,n=7)
이때
라고 하자. 위 상황을 기하학적으로 나타내면 다음과 같다.
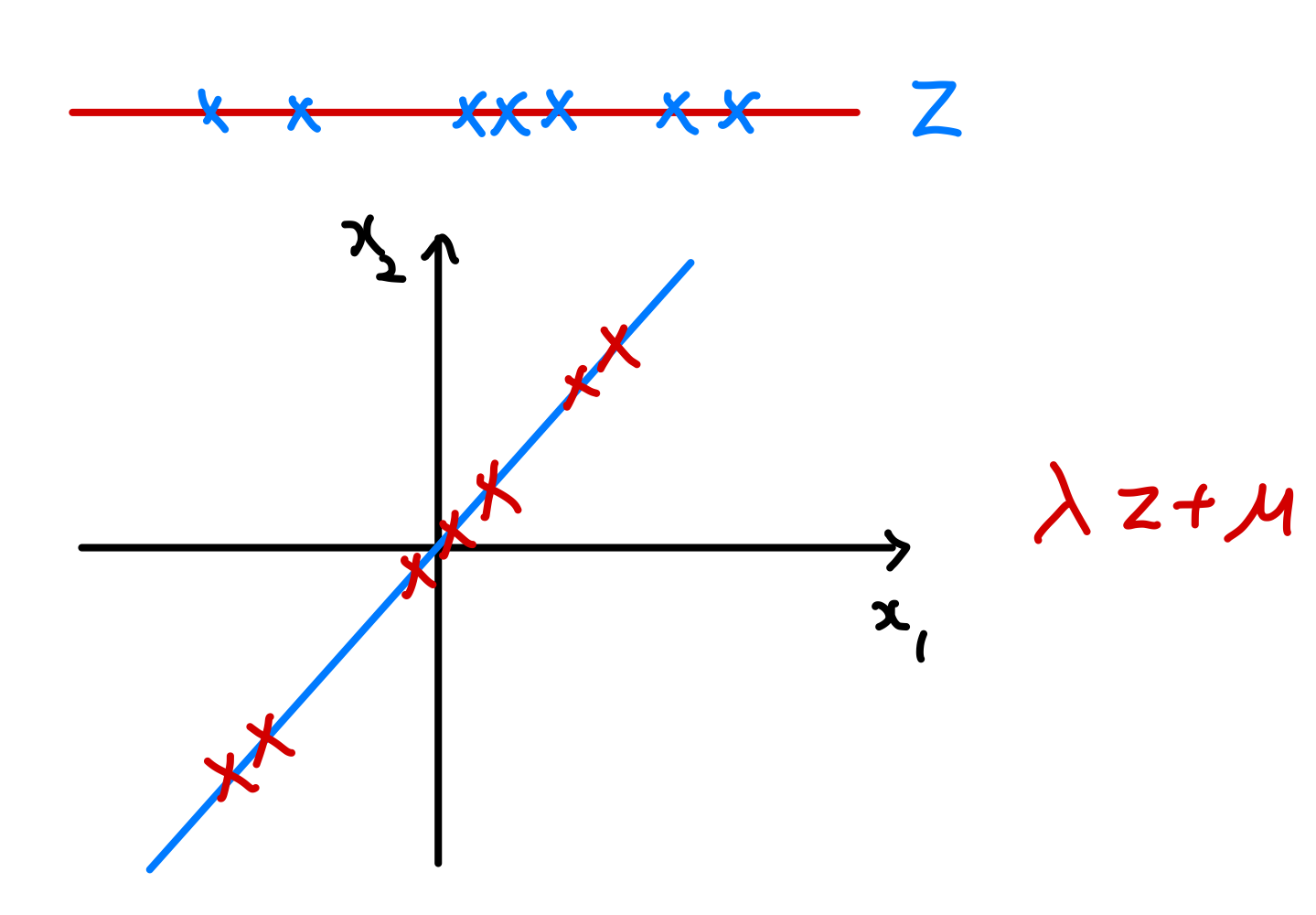
추가적으로 Ψ가 다음과 같다고 하자.
그러면 error에 대한 distribution은 다음과 같을 것이다.
(이때 가로는 x1, 세로는 x2를 의미하게 된다.)

error까지 더해준 결과는 다음과 같다.

이때 분홍색으로 표기한 점이 위의 model로부터 생성된 sample이다.
위 상황에서는 data는 2차원 공간안에 살고 있음에도 불구하고, 실질적으로는 굉장히 적은 차원에서 살고 있다. 따라서 그보다 작은 공간으로 projection시키되, 일종의 noise를 살짝 줌으로써 smoothing한 것으로 이해하면 된다.
Example 2
앞서 2개의 예시에서 살펴본 것처럼, factor-analysis를 사용하면 high-dimensional data를 활용할 수 있게 된다.
Marginals and Conditional of Gaussian Distribution
Suppose x∼N(μ,Σ) where
where x1∈Rr,x2∈Rs,x=Rr+s and
First, we want to compute the marginal distribution of x1(i.e p(x1)).
By the definition
So we can easily show that
Secondly, we want to compute the conditional distribution of x1∣x2
By the definition
So we can easily show that
정확한 증명은 다음 사이트를 참고하면 된다.
 https://statproofbook.github.io/P/mvn-cond.html
https://statproofbook.github.io/P/mvn-cond.htmlEM Algorithm for Factor Analysis
1. Derive p(x,z)
Since
where μ∈Rd,0∈Rk
Similarly, we can derive
Let's calculate one of the elements Σ22 in the above covariance matrix.
Since, z and ϵ are not correlated and their expectations are 0,
By doing similar calculations, we can easily find that
Finally, we can derive
2. E-step
Mixture of Gaussian의 경우에는 p(z(i)∣x(i);Θ)를 적당히 summation해서 구할 수 있었다. 하지만 여기서는 z가 continuous random variable이기 때문에, 위에서 증명한 conditional distribution of gaussian 공식을 활용해야 한다.
위 내용을 활용해서 Qi(z(i))를 표현한다.
3. M-step
M-step에서는 Qi(z(i))를 고정하고 다음 식을 maximize하고 싶은 상황이다.
여기서 하나의 트릭은 적분을 expectation 으로 돌릴 수 있다는 것이다. 즉 위의 식을 다음과 같이 바꿀 수 있다.
그러면 결과적으로 각 parameter에 대해서 영향을 주는 것은 ∑i=1nEz(i)∼Qi[logp(x(i)∣z(i);μ,Λ,Ψ)] 가 유일하다.
이때
위 식을 각각 μ,Λ,Ψ에 대해서 미분하고 0이 나오는 값을 찾아주면 된다. (Quadratic function임을 보장할 수 있으므로 미분하고 0인 값만 찾아줘도 된다.)상당히 복잡한 과정이 있지만 결과만 살펴보자면 다음과 같다.
이때
로 설정한다. 즉 Ψ가 Diagonal이 되게끔 하는 것이다.
정확한 유도는 다음을 참고하도록 하자.
Mixture of Factor Analysis
The assumption is that there are a number of different subpopulations (clusters), each of which is modeled with its own factor analysis model. This model is powerful because it can capture complex structures in the data. The factor analysis part allows it to capture correlations in the data, while the mixture model part allows it to represent multiple different subpopulations. This makes it particularly useful for things like clustering high-dimensional data, dimensionality reduction, and exploratory data analysis.
먼저 Factor analysis를 다시 확인해보도록 하자.
즉 결과적으로 x=μ+Λz+ϵ 의 형태로 표현이 된다는 것을 가정한 것이다.
반면, Mixture of Factor Analysis는 이와 달리 Factor Analysis들의 linear combination으로 p(x)가 표현된다고 가정한다
where
clustering(Mixture model) 과 dimensionality reduction(Factor analysis)를 동시에 수행하게 된다.
Nonlinear Dimensionality Reduction

만약 데이터가 다음과 같이 manifold상에 존재한다면, 단순한 linear pca만으로는 데이터들의 특성을 반영해주기 어렵다. 이러한 문제를 해결하고자 등장한 개념이 non-linear PCA이다.
하나의 더 예시를 살펴보도록 하자.

A better dimensionality reduction can be done by PCA after mapping of data points to feature space.
alternative representation of the original data that is obtained through some transformation or feature extraction process. It aims to capture the relevant information or patterns in a more suitable and informative way for analysis or modeling.즉 원래 공간에서 차원 축소가 잘 진행되지 않는 형태의 data distribution을 feature space로 transformation시키면 상대적으로 좋은 형태의 차원 축소를 진행시킬 수도 있다는 것
The objective for Kernel PCA
앞서 SVM with kernel에서 살펴본 것처럼, 계산적으로 cost가 크기 때문에 직접적으로 feature space에서 계산하는 것보다는 kernel function를 통해 계산하고자 하는 것이다.
앞서 PCA에서 했던 작업과 같이 feature space 상에서의 empirical covariance matrix를 구하면 다음과 같다.
where M : the dimension of feature space
논의의 편의를 위해 평균이 0이라고 가정을 하자. 그러면
그러면 m-dimensional principal subspace를 구성하고자 하는 경우에는 objective가 다음과 같다.
즉 S의 eigenvalue 중에 가장 큰 m개를 찾는 것이 목표이다.
이를 위해서는 일단 S의 eigenvector와 eigenvalue를 찾아야 한다. 그런데 식을 잘 관찰해보면 다음과 같은 특성을 확인할 수 있다.
where v is a eigenvector of S.
즉 다시 말해서 v는 {ϕ(xi)}i=1n들의 linear combination으로 표현할 수 있다는 의미이다.
So, there exist some coefficients {αn}n=1N such that
By using the above equation,
양변에 ϕ(xl)T를 곱한다
where α=[α1,…,αN]T and K2=KK (Matrix multiplication)
여태까지의 흐름을 요약하면 다음과 같다.
라고 했을 때
를 만족하게끔 하는 λi들을 찾고 싶은 것이다. 즉 S의 eigenvalue들 중 가장 큰 상위 m개를 택해주면되므로, 우리는 S의 eigenvector와 eigenvalue를 찾는 문제로 바뀌게 된다.
앞에서 증명한 것처럼 해당 문제는 결과적으로는 다음 식을 만족하는 α와 λ를 찾는 문제를 푸는 것과 동치가 된다.
이제 우리는 위 식을 더 정리하려고 한다.
일단, K가 symmetric positive semi-definiteness이다. 즉, 모든 eigenvalue가 non-negative이다. 따라서 K는 항상 eigen-decomposable하다. 이때, K의 eigenvector를 {wi}i=1N 이라고 하고, 이에 대응되는 eigenvalue를 {δi}i=1N이라고 하자. 이때, {wi}i=1N는 basis를 이루므로
를 만족하는 {ζi}i=1N이 존재한다. 따라서
따라서
Since w1,…,wN are independent
If δi=0,
이때 principal subspace를 구할 때 일반적으로 eigenvalue가 0인 값들에 대해서는 고려하지 않으므로 다음과 같은 식을 푸는 문제로 치환해서 풀 수 있게 된다.
이때 K∈L(N,N)이고 S∈L(M,M)이다. (M은 feature space의 dimension으로 N<M)
따라서 feature space 상에서 바로 eigenvalue decomposition을 수행하는 것보다 훨씬 computational적으로 훨씬 유리하다.
Normalization in Kernel PCA
사실 정확하게 하려면 v를 unit-vector로 잡아야 한다. 왜냐하면
라고 했을 때
결과적으로 우리가 원하는 값이 λ와 같아지기 때문이다. 따라서 앞의 연산에 추가적으로 v가 unit vector라는 constraint를 주어야 한다. 이에 따라 α에도 제한이 가해지게 된다.
Therefore,
즉 다음과 같은 λ를 찾는 작업으로 바뀌게 된다.
Compute Nonlinear Components
여태까지의 내용을 총 정리해보도록 하자. 또한 일반성을 잃지 않기 위해 principal subspace의 차원을 m으로 가정하겠다.
Suppose we have a dataset {xi}i=1N and this dataset is not easily linearly separable.
We want to increase the dimension by using feature mapping so that this dataset could be easily separable in the feature space. So, our objective is as follows
where
zeroSince we have some constraints for vi, we have to apply for Lagrange multipliers.
Differentiate it w.r.t vi
Therefore
is equivalent to
So, this problem is actually the problem of finding the eigenvalue of S.
Since S∈L(M,M), the computational cost for finding eigenvectors and eigenvalues is very large. We want to reduce the computational cost by using kernel trick
Actually, the problem which we actually want to solve is finding the solution of the following equation
We can convert this equation as follows
This means that vi can be expressed by the linear combination of {ϕ(xi)}i=1N. We say
By using this fact, we can convert the problem of Svi=λivi to
and multiply ϕ(xl)T each equation (i.e l is arbitrary)
where δ=[δ1,…,δN]T, K(i,j):= i, jth component of Kernel matrix
By using some calculations, the above problem could be converted to
In addition, by ∥vi∥=1
Finally, we want to solve this equation
Since δi and vi has 1-1 correspondence, if we know one of them, the other one can be easily calculated. In addition, if we find such δi (i.e. we know the eigenvalue of δi), we can easily calculate the λi which we actually want to find. As you expected, since K∈L(N,N), we can easily find the λi for O(N3) not O(M3).
So our objective is converted as follows
After we find such wi, this means that we actually know {vi}i=1m. In other words, we find the principal directions in the feature space. So how can we project new data into the subspace that we found?
Let’s say the subspace we found to E and the new data to x.
- We have to convert x to ϕ(x) (i.e transform data space to feature space)
- Project ϕ(x) to the subspace we found
where K is a Kernel function
Centering in Feature Space
기존의 Kernel matrix는 다음과 같다.
이때, feature space에 대응되는 값들의 평균을 0으로 만들고 싶은 상황으로 이해해주면 된다.
이를 기반으로 Kernel matrix를 다시 정의해주면 다음과 같다.
Therefore,
왜냐하면 1NK의 i,j 원소는 j번째 열의 평균, K1N의 i,j 원소는 i번째 행의 평균, 1NK1N은 전체 평균이기 때문이다.
이때, (I−1N)=(I−1N)−1이므로 K~는 K의 basis change에 대한 결과로 볼 수 있다. 즉 단순히 기저를 변경함으로써 feature space상의 데이터들의 평균을 원점으로 재조정한 것에 불과하다. 또한 K는 이미 eigenvalue decomposable하고 기저 변경에 의해서는 eigenvalue를 바꾸지 않으므로 최종적으로 우리의 objective를 다음과 같이 이해할 수 있다.
Isomap

Isomap approximates geodesic distance by using shortest paths in graph
즉 다시 말해서 2개의 data 사이의 metric를 graph 상에서 shortest path로 정의하겠다는 의미이다.
정확하게는 data 사이의 거리가 특정 임계값보다 적으면 edge를 그리고 해당 edge에 가중치를 부여하는 방식으로 그래프를 그린다. 그리고 해당 그래프에서 shortest path 문제를 푸는 것이다.
Isomap is a nonlinear dimensionality reduction technique that aims to preserve the global geometric structure of the data. It achieves this by first constructing a neighborhood graph based on pairwise distances between data points and then computing the shortest path distances (geodesic distances) on this graph.
Once the geodesic distances are computed, Isomap creates a new distance matrix that captures the intrinsic relationships between data points in the low-dimensional space. This new distance matrix is constructed by preserving the pairwise distances in the original high-dimensional space, while accounting for the manifold's underlying geometry.
To obtain the low-dimensional representation, Isomap performs eigen-decomposition on the new distance matrix. Eigen-decomposition is a process that decomposes a matrix into its eigenvectors and eigenvalues. In this context, Isomap computes the eigenvectors and eigenvalues of the new distance matrix.
The eigenvectors represent the principal components or basis vectors in the low-dimensional space, and the eigenvalues indicate the importance of each eigenvector in capturing the variance or structure of the data. By selecting a subset of the eigenvectors corresponding to the largest eigenvalues, Isomap obtains the low-dimensional representation of the data.
Autoencoder
앞서 살펴본 것처럼 kernel PCA나 isomap의 경우에는 각 데이터들에 대한 metric을 계산할 필요성이 존재했다. kernel PCA의 경우에는 feature space로 올라감에 따라 계산량이 올라가서 문제였고, isomap의 경우에는 geodesic distance를 구하는 것이 문제가 된다.

→ 따라서 metric을 정의하지 않고 알아서 해줬으면 좋겠다는 측면에서 등장한 개념이 Autoencoder 이다. 즉 data 그 자체로부터 mapping function을 배우게끔 하자는 것이 핵심 아이디어이다.
Encoder는 funciton이며 다음과 같이 정의된다.
즉 다시 말해서 data가 data들의 특성을 반영하고 있는 latent variable z에 대응되는 f를 찾고 싶은 것이다.
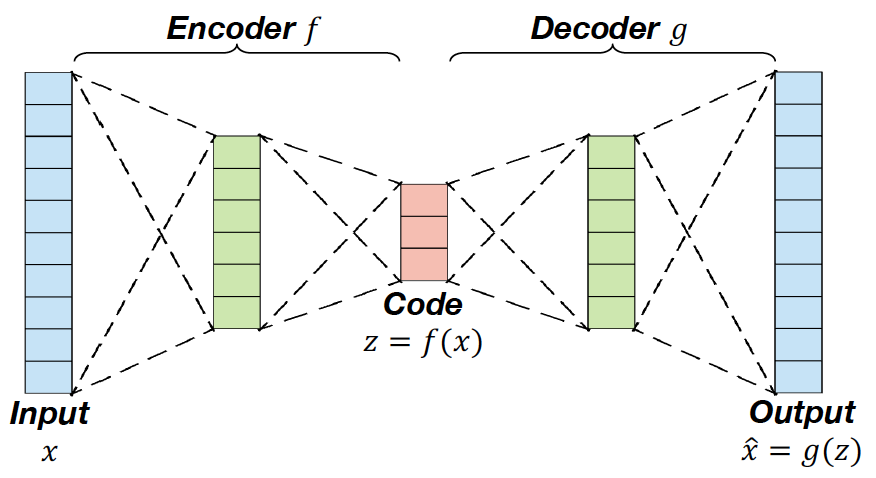
그래서 loss function은 다음과 같이 정의된다.
이때, encoder f와 decoder g는 parametric function이 될 수도 있다. 예를 들어 CNN이나 MLP가 그 예시이다.

예를 들어 이미지의 경우 f에 convolution operation을 g에 transposed-convolution operation을 취하는 것을 선택할 수 있다.
t-SNE
앞서 살펴본 것처럼, data compression 목적으로 dimensionality reduction을 수행할 때의 가장 큰 목적은 information preserving 이다. 즉 데이터의 정보 손실을 최소화하면서 차원을 축소하고 싶은 것이다.

하지만 t-SNE같은 visualization을 목적으로 dimensionality reduction을 수행할 때의 가장 큰 목적은 similarity preserving 이다.
Suppose we have a dataset {x1,…,xN}, we want to project a dataset to the lower dimension(R2 or R3).
이때, t-SNE의 목표는 data space 상에서의 거리(P)와 projection시킨 space상에서의 거리(Q)가 최대한 유사하게끔 하고 싶은 것이다. 단 data space상에서의 거리를 정의할 때 gaussian distribution을 사용하고 projection시킨 space상에서 거리는 t-distribution을 사용한 것이다.
이때
P와 Q의 차이가 작아지게끔 하는 것이 목표이므로
를 최소가 되게끔 gradient descent를 해주면 된다.
소중한 공감 감사합니다






